Isaac CHOTARD, Sindbad WALTER, Jomaï Hervé
Projet Brocoli
Activitée : Bourgeons de goût
Comment expliquer le fait que certaines personnes ne supportent vraiment pas le goût du brocoli?
Un Peu d'Histoire:
Dans les années 30, le chimiste Arthur Fox synthétise, le composé PTC (Phénylthiocarbamide), un composé amer produit entre par le brocoli. Il met en évidence à l’aide d'expérience que certaine personne était moins sensible que d’autre au brocoli et que cela était dû à un trait génétique récessif
Expérience n°1.1: La Perception de l'amertume
Comment expliquer le fait que certaines personnes ne supportent vraiment pas le goût du brocoli?
Chaque groupe dispose de 2 bandelettes, une ne contenant pas de PTC, le témoin négatif et l’autre imprégné de PTC, le test.
L’expérience se fait à deux, un testeur et un expérimentateur. Le testeur se fait bander les yeux et l’expérimentateur donne, sans préciser, successivement les 2 bandelettes, test et témoin à déposer sur sa langue. Puis les jette immédiatement après dans la javel, pour éviter les risques viraux.
Nous avons effectué ce test à trois reprises sur trois sujets différents, Isaac, Jomaï et Sindbad.
Isaac, a reconnu la bandelettes imprégné de PTC, son phénotype est donc [sensible]. Jomaï et Sindbad n’ont pas reconnu la bandelettes imprégné de PTC, leurs phénotype est donc [non-sensible].

Expérience n°1.2 : Les Bourgeons du Gôut
Nous disposons d’un système gustatif très développé qui nous permet de détecter et analyser avec précisions les cinq saveurs principales:
Sucrée - Salée - Acide - Unami - Amer
La saveur amère est la plus méconnue et la plus difficile à étudier à cause du grand nombre de composés amers existant.

Photographie d’une langue avec une loupe à main
A l'aide de la loupe à main nous avons mis en évidence les papilles gustatives présente sur la langue. Il y a quatre types de papilles, les trois premiers détectent les saveurs, le goût des aliments et le dernier détecte la forme et la consistance des aliments.
Cliquez sur l'image pour en savoir plus
Notre Hypothèse
A la suite de ces expériences nous pouvons nous demander ce qui affecte la sensibilité au PTC chez les individus. Nous avons plusieurs hypothèse à ce sujet mais la plus probable est la suivante. Nous pensons que cette différence de sensibilité est génétique. Le document 1 nous informe que l’incapacité à sentir le PTC est un trait génétique. De plus l’observation de la coupe transversale de langue de lapin nous pousse à penser que cette différence de sensibilité est dû à une mutation du gène codant la capacité à sentir le PTC. Différent allèle de ce gène permettent une complémentarité plus ou moins atteinte entre la molécule de PTC et le pore gustatif. Le pore gustatif pourrait donc être plus ou moins apte à acceuillire la molécule de PTC
Expérience n°2 : Les protéines, c'est la vie
Comment étudier les protéines ? Comment comprendre leur fonction physiologique ?
Introduction: Les acides aminés sont les éléments principaux constituant les protéines.
Une succession d’acides aminés assemblé grâce à des liaisons peptidiques permet de créer une protéine. Les acides aminés possèdent deux fonctions: amine avec la molécule (NH2) et acide carboxylique (COOH). Les différences qui permettent de les dissocier des uns des uns sont principalement les chaînes latérales (R) qui représente soit un assemblage d’atome ou par exemple un atome d’hydrogène. Dans le monde du vivant, il existe plus de 100 différents acides aminés mais seulement 20 sont utiliser pour codés le génome de chaque individu vivant. L'ordre des acides aminés et les différents enchaînement entre eux permettent de former les protéines spécifiques.
Les Acides Aminés
(quelques notions)
Acide Aminé (def) : Les acides aminés sont les éléments de base constituant les protéines. Ce sont des acides organiques contenant au moins un radical amine (NH2) et un radical carboxyle (CO2H).
Protéine (def) : Grosse molécule complexe d'acides aminés, constituant essentiel des matières organiques et des êtres vivants.
Structure Primaire (def) : Ordre des acides aminés le long de la chaîne polypeptidique.
Liaison Peptidique (def) : formation d’une liaison covalente entre le COOH d’un Acide Aminé et le NH2 d’un autre Acide Aminé par la perte d’une molécule d’eau.


Acides Aminés Apolaires




Noir : Carbone (C)
Bleu : Azote (N)
Blanc : Hydrogène (H)
Jaune : Souffre (S)
Rouge : Oxygène (O)
Acides Aminés Polaires




Noir : Carbone (C)
Bleu : Azote (N)
Blanc : Hydrogène (H)
Jaune : Souffre (S)
Rouge : Oxygène (O)
Acides Aminés Basiques (chargés positivement)




Noir : Carbone (C)
Bleu : Azote (N)
Blanc : Hydrogène (H)
Jaune : Souffre (S)
Rouge : Oxygène (O)
Acides Aminés Acidiques (chargés négativement)
.png)
.png)
.png)
Noir : Carbone (C)
Bleu : Azote (N)
Blanc : Hydrogène (H)
Jaune : Souffre (S)
Rouge : Oxygène (O)
Tableau comparatif des protéines

Expérience n°3 : Comprendre la structure 3D des protéines
Comment les interactions chimiques permettent de comprendre la forme des protéines (insuline et récepteur au PTC) ?
Rappels:
La structure d’une protéine est la formation d’une enchaînement d’acides aminés et la structure en trois dimensions de la protéine. Elle permet de connaître la position entre les atomes et la forme dans l’espace de la protéine formée.
Il existe 4 structures pour une protéine. Elle commence par une structure primaire et change d’une structure secondaire à tertiaire pour donner la version la plus complexe avec une structure quaternaire.
-
Liaison peptidique: Une liaison peptidique formé pendant l’étape de la traduction (cf. TP4) elle est créée quand la partie amine A (NH2)et la partie acide A (COOH)se lient par liaison covalente d’où “lien peptidique”.
-
Chaîne polypeptidique: Une chaîne sur laquelle est placée plusieurs acide aminés liées entre eux par liaison peptidique.
Nous avons étudier à l’aide d’Uniprot la structure de l’insuline. L'insuline est une hormone polypeptidique aide à maintenir le niveau de glucose dans le sang. Cette hormone est utilisée la plupart du temps pour aider des patients atteint de diabète.


Étude des différentes structures de l’insuline


L’hélice alpha est une des structures secondaires. Elle est crée lorsque l’hélice formé d’une chaîne polypeptidique enroulés sur elle-même dans laquelle chaque principale d’un acide aminé forme une liaison hydrogène entre les groupes (N-H) et (C=O).

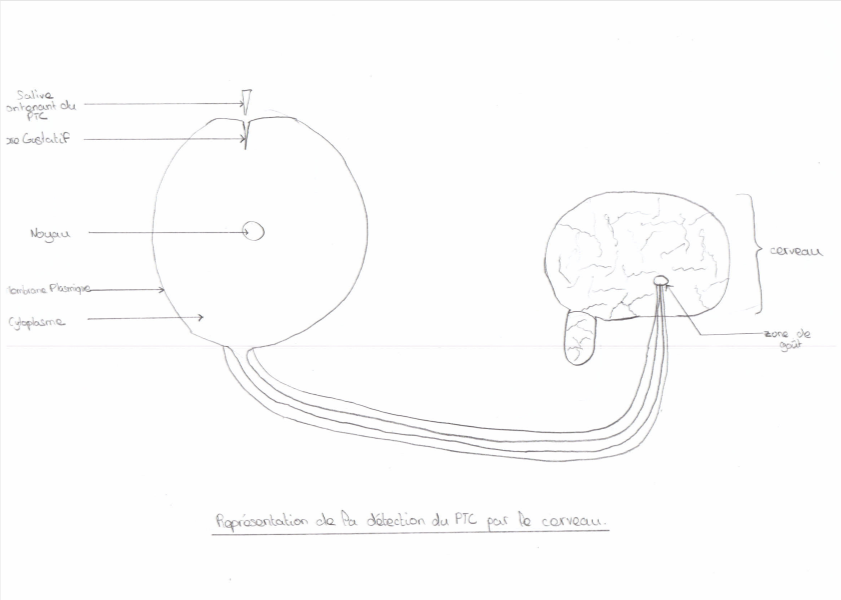
Schéma bilan d’une cellule du goût pour un individu sensible et non-sensible au PTC
Dans un premier temps, nous avons réalisé un schéma détaillé d’une cellule du bourgeon du goût pour un individu sensible au PTC. Le schéma observe deux phénotypes: microscopique et moléculaire. Dans ce schéma, nous observons que le PTC va interagir avec les deux acides aminés: Alanine 262 et Tyrosine 199 (représenté sous forme d’un ensemble de petites sphères respectivement en rouge et en vert). Le PTC va donc se placer sur ces endroits précis ce qui expliquerait la sensibilité au PTC d’un individu.

Dans un deuxième temps, nousavons réalisé un schéma détaillé d’une cellule du bourgeon du goût pour un individu non-sensible au PTC. Les deux acides aminés représentés en rouge et en vert dans le schéma précédent sont maintenant en bleu. On les a représentés ainsi, pour montré qu’elles ont subient des mutations ce qui pourrait expliquer la non-sensibilté au PTC d’un individu.

Séquence de l'Insuline
Certaines acides aminés favorisent la formation d’hélices . Les principaux sont : la méthionine (M), l'alanine (A), la leucine (L), le glutamate (E) et la lysine (K). Pour les retrouver dans la séquence d’acides aminés nous utilisons le code à une lettre des acides aminés (“MALEK”) que l’on cherche dans la séquence de la protéine pour prouver l’existence d’hélice alpha au sein de la protéine étudiée.
Grâce à Uniprot, on a regardé la séquence de l’insuline (lien actif P01308). En utilisant le format FASTA on a cherché s’il y avait une formation d’hélice grâce au code (“MALEK”).
MALWMRLLPLLALLALWGPDPAAAFVNQHLCGSHLVEALYLVCGERGFFYTPKTRREAEDLQVGQVELGGGPGAGSLQPLALEGSLQKRGIVEQCCTSICSLYQLENYCN
Nous constatons la présence de “MALEK” et donc la présence des acides aminés qui favorisent la formation d’hélice alpha. On conclut qu’il y a la présence d’au moins une hélice alpha.
Formation de deux ponts de disulfure:
Comme on le sait déjà il existe plusieurs catégories pour les acides aminés et on sait que les deux seuls acides aminés contenant du soufre sont la Méthionine (M, Met) et la Cystéine (C, Cys).
De plus, les acides aminés Cystéines peuvent se lier entre eux et créer une pont de disulfure car les deux molécules de souffres contenus dans chacune des acides aminés se lient, ainsi formant un pont solide dans l’espace.

Structure de la protéine représenté en ruban:
Dans une deuxième partie, à l’aide de Libmol, nous avons visualiser le récepteur nommé “TAS2R38” sensible au PTC et étudier les différentes structures de la protéine.




Étude de la structure tertiaire en coloration par chaîne bleue:
Fixation du PTC sur son récepteur protéique:
Le PTC se fixe sur son récepteur protéique à un endroit très précis: entre les hélices alpha TransMembranaires 3, 5 et 6. Le PTC fabriquerait des liaisons hydrogènes avec l’Alanine 262 du TM6 (correspond à l’acide aminé Ala 262 situé sur l’hélice TransMembranaires n°6) ainsi qu’avec la Tyrosine 199 du TM5 (correspond à l’acide aminé Tyr situé sur l’hélice TransMembranaires n°5). Grâce à ces liaisons fabriqué, la structure du récepteur est légèrement modifié et cela provoquerait l’activation du PTC.

Expérience n°4 : Les protéines, c'est codé par l'ADN
Comment l’ADN code-t-il pour une protéine ?
Passage de l’ADN à la protéine:
Les molécules d’ADN et ARN ont presque la même structure chimique. La seule différence notable est que la thymine (T) de l’ADN est remplacé par de l’uracile (U) dans l’ARN. Au niveau structure, la différence entre l’ADN et ARN est que ARN est monobrin. Pendant la division cellulaire, l’ADN vient couper l’ADN en deux brins qui forment un brin non-transcrit et un brin transcrit. Durant la transcription, l’ARN vient compléter le brin d’ADN transcrit qui se transforme en ARN messager. L'ARNm va donc sortir du noyau et le ribosome va ensuite scanner la succession d’acides aminés et associer chaque triplet de nucléotides à un acide aminé. La seule condition pour commencer la traduction est de trouver la méthionine avec une séquence AUG.
Mutation dans la séquence nucléotidique, que ce passe-t-il?
Si une mutation s’opère, une nucléotide va être enlevé (mutation par substitution) ou ajouté (mutation par insertion) au sein de la séquence du brin non-transcrit de l’ADN. Cette mutation va totalement changer la molécule à travers les différentes échelles. En premier, si il y a une mutation dans la séquence nucléotidique alors il y aura différents acides aminés. Cela provoque ensuite un changement au niveau de la protéine formé initialement. La protéine mutée va ensuite modifié la structure primaire, secondaire, tertiaire et quaternaire qui va finalement transformer la structure de la protéine.

Durant le 1er TP on a observé que la sensibilité au PTC était peut-être lié à la structure du PTC. Comme on le sait chaque protéine à une forme spécifique et il y a beaucoup de combinaison possible. Le PTC en revanche à une structure spécifique et si on n’a pas “la bonne clé pour la serrure” alors le PTC ne laisse pas rentrer la protéine car elle ne s'emboîte pas avec la forme du PTC.
Donc si une mutation s’opère à une échelle minuscule (10 puissance -10) alors durant la synthèse protéique tout bascule. Au final, la structure de la protéine initial est modifié ce qui résulte à une non-complémentarité entre la structure du PTC et la protéine.
Les differentes molécules du vivant
Nous avons construit des maquettes, pour les quatres molécules du vivant (Adénine; Cytosine; Guanine; Thymine).
Grâce à ces maquettes nous avons put remarquer pourquoi chaque molécule ne pouvait former une liaison hydrogène (atome + hydrogène + atome) avec qu'une seule autre molécule.
(ex: Adénine + Thymine ou Cytosine + Guanine)

Ci-contre, les molécules de Thymine et Guanine ne se lient pas ensemble.
(O - O et C - H - H - N)

Ci-contre, les molécules de Thymine et Cytosine ne se lient pas ensemble.
(N - H - N et N - O)

Ci-contre, les molécules de Thymine et Adénine se lient ensemble.
(N - H - C et O - H - N)

Ci-contre, les molécules de Adénine et Guanine ne se lient pas ensemble.
(C - H - H - N et N - H - N et N - H - O)

Ci-contre, les molécules de Cytosine et Guanine se lient ensemble.
(O - H - N et N - H - N et N - H - O)

Ci-contre, les molécules de Adénine et Cytosine ne se lient pas ensemble.
(N - H - H - C et N - N et O - H - N)
Expérience n°5 :
Problème de Fermi :
1) On sait que le volume de l’échantillon prélevé est de 3,0 x 10^-6 L et qu’après un cycle de multiplication le volume se multiplie par deux. Or il y a trente cycles de multiplication donc il faut multiplier le volume de l’échantillon par 2^30.
3,0 x 10^-6 x 2^30 = 3 221,2 L
Piscine Olympique : 50 x 3 x 10 = 1 500 000 dm^3 (L)
Citerne d’eau : 3500 L
Becher : 0,250 L
Bouteille : 1 L
Sceau : 15 L
Baignoire : 150 L
2) En prenant comme échantillon la classe de 1S4 nous pouvons constater que sur 26 élèves 4 n’aime pas le brocoli et donc sont probablement sensible au PTC. Ainsi, seulement 15% de l’échantillon est sensible au PTC.
Liaison hydrogène dans la liaison entre les nucléotides Adénine et Thymine et énergie pour les casser:
-
N-H---O : 10 kJ.mol-1
-
N-H---N : 25 kJ.mol-1
Energie nécessaire pour casser une liaisons Adénine-Thymine :
-
10 + 25 = 35 kJ.mol-1
Liaison hydrogène dans la liaison entre les nucléotides Cytosine et Guanine et énergie pour les casser:
-
2 N-H---O : 10 kJ.mol-1
-
N-H---N : 25 kJ.mol-1
Energie nécessaire pour casser une liaisons Cytosine-Guanine:
-
10 * 2 + 25 = 45 kJ.mol-1
Pour un individu au phénotype sensible au PTC:
-
Nombre de liaisons Adénine-Thymine :
-
201 + 307 = 508
-
-
Nombre de liaisons liaisons Cytosine-Guanine :
-
265 + 229 = 494
-
Energie nécessaire afin de casser toutes les liaisons dans la séquence ADN d’un individu au phénotype sensible au PTC:
-
(508 * 35) + (494 * 45) = 4,00 * 10^4 kJ.mol-1
Pour un individu au phénotype non sensible au PTC:
-
Nombre de liaisons Adénine-Thymine :
-
201 + 308 = 509
-
-
Nombre de liaisons liaisons Cytosine-Guanine :
-
263 + 230 = 493
-
Energie nécessaire afin de casser toutes les liaisons dans la séquence ADN d’un individu au phénotype non sensible au PTC:
(509 * 35) + (493 * 45) = 4,00 * 10^4 kJ.mol-1
Nous observons que l’énergie pour casser toutes les liaisons dans la séquence ADN d’un individu ne varie pas selon si il a un phénotype sensible ou non au PTC.
Pour tous les individus, l’énergie nécessaire est de :
-
4,00 * 10^4 kJ.mol-1
Calculons la quantité de mol d’ADN dans notre échantillon.
n(ADN) = (ρ(ADN) * V(ADN)) / M(ADN) = (1,6 * 3,0 * 10^-3) / (9,9 * 10^11)
n(ADN) = 4,8 * 10^-15 mol
A l’aide d’une règle de trois calculons la quantité d’énergie nécessaire pour casser la quantité d’ADN de l'échantillon :
4,8 * 10^-15 * 4,00 * 10^4 = 1,92 * 10^-10 kJ
Le nombre de cycle de la PCR est de 30, donc la quantité d’énergie utilisé est de
1,92 * 10^-10 * 2^30 = 2,06 * 10^-1 kJ
2,06 * 10^-1 kJ = 2,06 * 10^2 J
Or 1 cal = 4,18 J
Donc 2,06 * 10^2 J = 4,93 * 10^1 cal
Soit t, le temps en heure de la course à pied de référence utilisé dans les données.
Un joggeur de 60 kg dépensera 738 cal s'il cours à une vitesse de 12 km/h pendant t heures.
Il mettra donc 6,68 * 10^-2 * t heures pour dépenser la même quantité d’énergie que la TAQ polymérase durant ses 30 cycles.
Un joggeur de 70 kg dépensera 880 cal s'il cours à une vitesse de 12 km/h pendant t heures.
Il mettra donc 5,60 * 10^-2 * t heures pour dépenser la même quantité d’énergie que la TAQ polymérase durant ses 30 cycles.
Un joggeur de 80 kg dépensera 1078 s'il cours à une vitesse de 12 km/h pendant t heures.
Il mettra donc 4,57 * 10^-2 * t heures pour dépenser la même quantité d’énergie que la TAQ polymérase durant ses 30 cycles.
Supposons que la course de référence a duré 1 heure.
Le joggeur de 60 kg mettra donc 6,68 * 10^-2 heures soit 4,01 minutes.
Le joggeur de 70 kg mettra donc 5,60 * 10^-2 heures soit 3,36 minutes.
Le joggeur de 80 kg mettra donc 4,57* 10^-2 heures soit 2,74 minutes.
Supposons que la masse moyenne d’un humain soit de 70 kg, un humain mettrai donc environ 3,36 minutes pour dépenser la même quantité d’énergie que la TAQ polymérase durant ses 30 cycles.
Expérience n°6 (PC) : PCR et Électrophorèse
Définition Champs :
Un champs est une perturbation d’un espace donné qui, dans chaque point, est
due à une grandeur physique indépendante des corps qui peuvent s'y trouver, cette grandeur pouvant être un nombre (température, pression, …) ou un vecteur (vitesse, champ magnétique dû à un aimant …).
« Le champ crée une toile à travers tout le ciel ».
Citation de Maxwell (fondateur de la notion de champ)
Son effet peut être gravitationnel lorsqu’il est lié à la Terre, électrique autour d'une
charge, ou magnétique autour d'un courant électrique ou d’un aimant.




L’électrophorèse (def) : L’électrophorèse est une opération entre deux armatures, dans ce cas-là des électrodes (l’un est chargé positivement et l’autre est chargé négativement). Le champ électrique créé entre ces deux électrodes est uniforme(cf Notion de champ). La Seconde Loi de Newton nous explique donc que les échantillons plus lourd se déplaceront plus lentement que les échantillons plus léger.

Expérience n°6 (SVT) : PCR et Électrophorèse
Comment déterminer si un individu est homozygote ou hétérozygote pour le gène TAS2R98?
Matériel utilisé :

Centrifugeuse : Lorsque nous plaçons les tubes à PCR dans la centrifugeuse, elle se met à tourner à 5000 tours minutes afin de plaquer la solution au fond des tubes et de l'homogénéiser.

Mini PCR (8 trous) : La mini PCR permet de chauffer les tubes à PCR pendant un durée déterminée à l’avance de manière très précise.

Cuve à Électrophorèse : cuve permettant de déplacer les brins d'ADN grâce à des charges électriques.
Le protocole expérimental du TP est séparé en quatre phases:
Étape A - Extraction ADN des cellules buccales :
Dans un premier temps, nous avons choisi deux individus pour qui,
d’après la première expérience (TP1), nous avons pu conclure qu’ils étaient sensibles et insensibles au PTC. Les deux sujets ont d’abord gratté, à l’aide d’un cure-dent, l’intérieur de leurs joues en répétant cette action six fois. Ce processus permet donc de récupérer les cellules buccales de l’individu.
Dans un deuxième temps, nous avons écrasé le cure-dent à
l’intérieur d’un tube à PCR contenant 50 µl de l’Xtract Buffer (qui sert à ouvrir la membrane des cellules et extraire l’ADN du noyau) pour que les cellules buccales se détachent du cure-dent et s’installent au fond du tube à PCR.
Dans un troisième temps nous avons centrifugé la solution afin de
l’homogénéiser, enlever les bulles d’air et ramener l’ADN au fond du tube. Dans un dernier temps, à l’aide du Mini PCR nous avons pu chauffer la solution à 95°C pendants 10 minutes.
Étape A - Extraction ADN des cellules buccales :
Dans un premier temps, nous avons choisi deux individus pour qui,
d’après la première expérience (TP1), nous avons pu conclure qu’ils étaient sensibles et insensibles au PTC. Les deux sujets ont d’abord gratté, à l’aide d’un cure-dent, l’intérieur de leurs joues en répétant cette action six fois. Ce processus permet donc de récupérer les cellules buccales de l’individu.
Dans un deuxième temps, nous avons écrasé le cure-dent à
l’intérieur d’un tube à PCR contenant 50 µl de l’Xtract Buffer (qui sert à ouvrir la membrane des cellules et extraire l’ADN du noyau) pour que les cellules buccales se détachent du cure-dent et s’installent au fond du tube à PCR.
Dans un troisième temps nous avons centrifugé la solution afin de
l’homogénéiser, enlever les bulles d’air et ramener l’ADN au fond du tube. Dans un dernier temps, à l’aide du Mini PCR nous avons pu chauffer la solution à 95°C pendants 10 minutes.


Etape B - Amplification de l’ADN par PCR :
Pour commencer, nous avons extrait 3 µl de la solution d’ADN
conçue dans l’étape A pour l'insérer dans un nouveau tube à PCR. On y ajoute 13 µl de PTC primer : l’amorce, permettant de mettre des bornes autour de la séquence d’ADN qui nous intéresse, plus particulièrement le gène TAS2R98 qui codent pour la protéine réceptrice du PTC. Ensuite, on y ajoute 13 µl d’EZ PCR master mix contenant un mélange de nucléotides complémentaires au brin d’ADN et de la Taq polymérase permettant de recopier et d’amplifier l’ADN.
Après, nous avons inséré le tube contenant le mélange dans les
trous de la mini PCR. Ensuite, grâce au logiciel de l’ordinateur nous avons modifié les paramètres. Nous avons demandé au logiciel de faire 30 cycles de PCR comprenant: 3 “phases”. Cette étape dure 90 minutes.
Processus au cours d’un cycle : au cours de la première phase c’est
la dénaturation nous chauffons les tubes de PCR donc l’ADN à 94°C pendant 10 secondes afin d’agiter l’ADN et détruire les liaisons hydrogènes entre les deux brins d’ADN. Après cette séparation, la deuxième phase se met en place: “annealing” ou “hybridation” qui permet grâce au PTC primer d’accrocher les deux amorces aux endroits nécessaires tout cela bien sûr ce fait dans une température à 58°C (car à 95° il y a trop de mouvements) pendant 15 secondes. Finalement, la dernière étape : extension permet de recopier l’intégralité du gène à 72°c pendant 40 secondes
Etape C - Digestion de l’ADN par les enzymes de restriction :
Nous avons placé 14 μL de l’ADN après PCR dans un tube à PCR à l’aide
d’une micropipette pouvant contenir entre 10μl-100μl. Puis, à l’aide d’une micropipette pouvant contenir entre 1 μl et 10 μL nous avons placé 1 μl de l’enzyme de restriction Fnu 4 HI dans le même tube à PCR. Afin d'homogénéiser la solution et d’enlever les bulles nous avons utilisé une centrifugeuse (cf étape B). Puis nous avons chauffé les tubes à PCR à 37°C pendants 15 minutes afin de faire agir les enzymes.


Interprétation des résultats de l’électrophorèse:
L’étalon est composé de 5 fragments allant de 100 à 500 paires de bases. Ces fragments vont nous aider à confirmer la taille des fragments digéré.
Il y a trois possibilitées pour les allèles du gène PTC. La première est d’avoir deux allèles codant pour un récepteur sensible au PTC, l’individu est donc homozygote sensible.

La seconde est d’avoir deux allèles codant pour un récepteur non-sensible au PTC, l’individu est donc homozygote non-sensible.

La troisième possibilitées est d’avoir un allèle codant pour un récepteur sensible et un pour un récepteur non-sensible, l’individu est donc hétérozygote.

Si un individu possède l’allèle codant pour un récepteur sensible au PTC,
l’enzyme va couper l’ADN une fois, créant donc deux fragments.
Si un individu possède l’allèle codant pour un récepteur non-sensible au PTC,
l’enzyme ne va pas couper l’ADN. Il n’y aura donc qu’un fragment.
Nous observons que l’ADN placé dans le puits numéro 2 est composées de
trois fragments donc l’individu est hétérozygote. Or nous savons que si un individu est hétérozygote, les fragments d’ADN seront de taille 250-150-100 paires de bases. Les fragments étalons permettent de confirmer ce résultat. Le test PTC ayant conjecturé un individu assez sensible c’est donc révéler véridique.
Nous observons que l’ADN placé dans le puits numéro 3 a un fragment,
l’individu est donc homozygote non-sensible. Or nous savons que si un individu est homozygote non-sensible le fragment d’ADN seront de 250 paires de bases. Les fragments étalons permettent de confirmer ce résultat. Le test PTC ayant conjecturé un individu non-sensible c’est donc révéler véridique.
Nous observons que l’ADN placé dans le puits numéro 4 a deux fragments,
l’individu est donc homozygote sensible. Or nous savons que si un individu est homozygote sensible, les fragments d’ADN seront de taille 150-100 paires de bases. Les fragments étalons permettent de confirmer ce résultat. Le test PTC ayant conjecturé un individu très sensible c’est donc révéler véridique.
Nous observons que l’ADN placé dans le puits numéro 5 a trois fragments,
l’individu est donc hétérozygote. Les fragments sont donc de taille 250-150-100. Les fragments étalons permettent de confirmer ce résultat. Le PTC ayant conjecturé un individu assez sensible c’est donc révéler véridique.

Contactez-nous
J'espère que vous avez aimé notre project, si vous avez un question ou vous souhaitez nous contacter, veuillez remplir ce formulaire :
